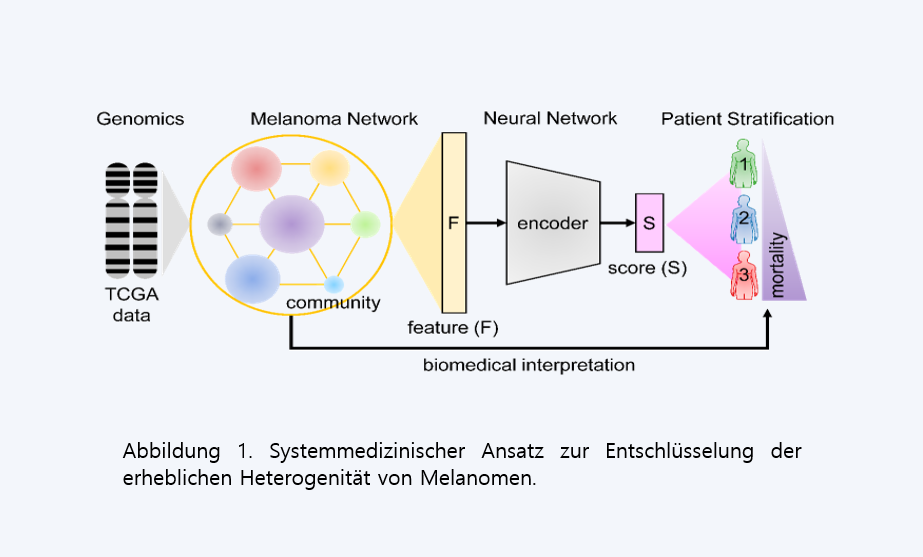
Beim kutanen Melanom treten multiple Genomveränderungen auf, von denen einige die Prognose beeinflussen können. Daher kann die Integration von Genomdaten und klinischen Ergebnissen die Identifizierung der für die Melanomprogression relevantesten Genommerkmale ermöglichen. Das Team aus der Hautlink des Universitätsklinikums Erlangen hat einen systemmedizinischen Ansatz entwickelt, der Genomdaten mit einem Krankheitsnetzwerk und einem Deep-Learning-Modell für die prognostische Klassifizierung von Melanompatienten integriert und die Einflüsse genomischer Merkmale analysiert (Abbildung 1). Das Deep-Learning-Modell nutzt insbesondere die im Netzwerk identifizierten Cluster („communities“), um die Dimensionalität der Genomdaten effektiv zu reduzieren und ein Patientenprofil zu erstellen. Anhand dieses Profils hat das Team drei Melanomsubtypen mit unterschiedlichen Überlebenszeiten identifiziert. Anschließend quantifizierten und bewerteten sie die Auswirkungen genomischer Merkmale auf das Patientenprofil mit Hilfe eines maschinellen Lernverfahrens. Die Analyse der am höchsten eingestuften Merkmale lieferte biologische Anknüpfungspunkte sowohl auf der Ebene der Signalwege als auch auf molekularer Ebene, z. B. die entsprechenden Mutations- und Interaktoms-Profile beim Melanom und die Beteiligung der Signaltransduktion, der Immunantwort und des Zellzyklus. Insgesamt hat die Leistungsfähigkeit netzwerkbasierter künstlicher Intelligenz bei der personalisierten Prognoseerstellung von Melanompatienten demonstriert. Der generische Charakter des Ansatzes legt nahe, dass er auch auf andere Krebsarten anwendbar ist.
Lai X, Zhou JF, Wessely A, Heppt M, Maier A, Berking C, Vera J, Zhang L. A disease network-based deep learning approach for characterizing melanoma. International Journal of Cancer. 2022; 150(6): 1029-1044. doi:10.1002/IJC.33860